메틸화 시퀀싱
메틸레이션 시퀀싱의 이점
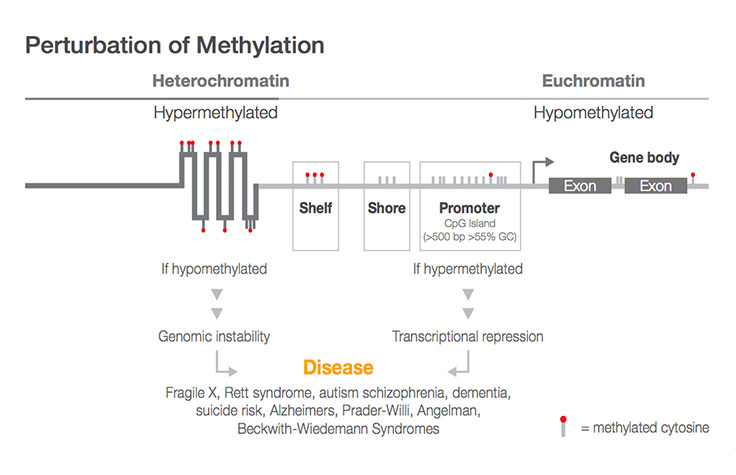
사이토신 메틸레이션은 시간적/공간적 유전자 발현과 크로마틴 리모델링을 크게 수정할 수 있습니다. 차세대 시퀀싱(next-generation sequencing, NGS)의 힘을 활용하는 전장 유전체 분석과 표적 메틸화 시퀀싱 접근법 모두 단일 뉴클레오티드 수준에서 메틸레이션 패턴(methylation pattern)에 대한 통찰력을 제공할 수 있습니다.
메틸레이션 시퀀싱의 장점:
- 인간 유전체 전체에서 CpG, CHH 및 CHG 영역의 메틸레이션 패턴 발견
- 전장 유전체 접근법인 전장 유전체 바이설파이트 시퀀싱(WGBS)을 통해 대부분의 종 전반에서 유전체의 거의 모든 사이토신에서 메틸레이션 확인
- 소량의 DNA로 전체 샘플의 다양성 캡처
- ENCODE, FANTOM5 및 Epigenomics RoadMap 컨소시엄에서 식별된 인간 유전체의 새로운 관심 영역을 표적 메틸화 시퀀싱으로 커버하세요
Advancing DNA Methylation Studies with NGS
이 eBook은 유전자 발현 및 조절 연구에 대해 설명합니다. 과학자들이 메틸레이션 시퀀싱 및 어레이(array) 방법을 사용하여 연구를 가속화하는 방법에 대해 알아보세요.
지금 다운로드메틸화 시퀀싱은 어떻게 작용합니까?
많은 접근법이 메틸레이션 분석을 위해 NGS의 높은 품질과 민감도를 활용합니다. 대부분의 방법은 메틸레이션되지 않은 사이토신을 검출하기 위해 DNA의 바이설파이트 전환에 의존합니다. 바이설파이트 전환은 라이브러리 준비 중에 메틸레이션되지 않은 사이토신을 우라실로 변경합니다. 전환된 염기는 시퀀싱 데이터에서 티민으로 식별되고(PCR 후), 판독 계수를 사용하여 메틸레이션 사이토신 %를 결정합니다.
바이설파이트 전환 시퀀싱은 앰플리콘 methyl-seq 또는 표적 인리치먼트와 같은 표적 방법 또는 전장 유전체 바이설파이트 시퀀싱을 사용하여 수행할 수 있습니다. 또한 OxBS 및 TAB-Seq와 같은 대체 화학은 메틸레이션(5-mc) 분석과 함께 하이드록시메틸레이션(5-hMc)을 식별하기 위해 NGS와 함께 사용할 수 있습니다.
메틸레이션 분석 방법에 대한 자세한 내용은 DNA 메틸레이션 분석에 대한 필드 가이드를 참조하세요.

멀티오믹스 통찰력을 얻기 위한 5-base 분석으로 복잡한 경로 디코딩
이 eBook에서, 유전 질환, 암 및 기타 복합 질환의 메커니즘을 파악하기 위해 유전자 변이와 DNA 메틸화를 함께 시퀀싱하는 것의 가치를 살펴보세요.
eBook 다운로드메틸화 연구 논문

Epigenetic Contributions to Addiction
Marilee Morgan은 물질 남용자의 뇌 영상 스캔과 후성유전학적 유전자 시그니처를 연관시키기 위해 표적 메틸레이션 시퀀싱을 사용하는 방법에 대해 논의합니다.

메틸화 및 유전자 변이체 시퀀싱
메틸화 연구를 위한 Illumina 5-base 솔루션은 메틸화 및 유전자 변이체의 동시 시퀀싱을 지원합니다.
유전체 및 메틸롬 시퀀싱에 대해 더 알아보세요.주요 메틸화 시퀀싱 워크플로우
라이브러리 준비에서 데이터 분석에 이르기까지 메틸화 시퀀싱을 위한 포괄적인 워크플로우에 액세스합니다. 전장 유전체 바이설파이트 시퀀싱은 모든 사이토신에 대한 염기 수준 메틸화 정량화를 제공합니다.
메틸화 시퀀싱 데이터 분석 도구
DRAGEN 2차 분석은 메틸화 시퀀싱 데이터 분석부터 전장 유전체, 엑솜 및 단일세포 RNA-Seq 데이터 분석에 이르기까지 광범위한 애플리케이션을 지원합니다.
DRAGEN에 대해 더 알아보기
주요 리소스
Infinium 메틸레이션 마이크로어레이 가이드
Infinium 메틸레이션 마이크로어레이 가이드로 후성유전학 연구를 강화하세요. 복잡한 생물학적 상호작용을 발견하고 중개 연구를 발전시키세요.
Advancing DNA Methylation Studies with NGS
이 eBook은 유전자 발현 및 조절 연구에 대해 설명합니다. 과학자들이 메틸레이션 시퀀싱 및 어레이(array) 방법을 사용하여 연구를 가속화하는 방법에 대해 알아보세요.
시퀀싱 방법 가이드
유전체, 전사체, 후성유전체 연구에 필요한 모든 것을 한 곳에서 찾아보세요. 종합적인 연구 가이드를 통해 랩에 가장 적합한 도구를 선택하세요.
관련 솔루션
후성유전학과 암
비정상적 메틸화, 변경된 전사 인자 결합 및 기타 후성유전적 변경에 대한 연구는 중요한 종양 형성 경로에 대한 인사이트를 제공할 수 있습니다. 암 후성유전학 연구에 대해 자세히 알아보세요.
복합 질환 유전체학
전장 유전체의 메틸레이션 시퀀싱은 연구진이 복잡한 신경학적, 면역학적 질병과 기타 질병에서 작용하는 기능적 메커니즘을 이해하는 데 도움이 될 수 있습니다. 복합 질환 연구에 대해 자세히 알아보세요.
소식지, 사례 연구 및 유전체 분석 기법에 대한 정보를 받고 싶으신가요?
이메일 주소를 입력해 주세요.